Descrizione
Obiettivi e risultati attesi
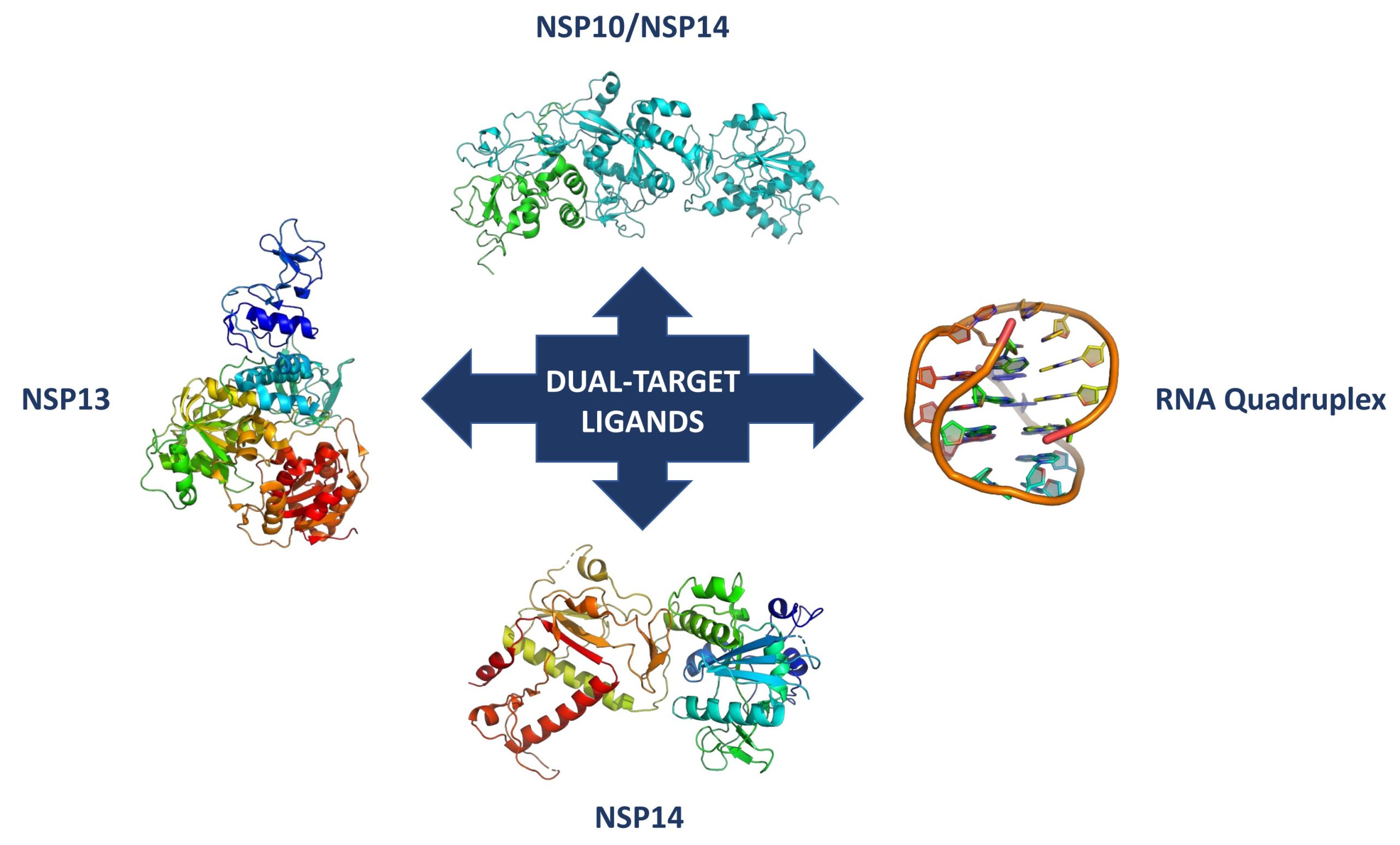
Il progetto MUTASAR sarà realizzato utilizzando un approccio multidisciplinare, che comprende chimica medica e computazionale, biologia molecolare, biologia strutturale, chimica biologica e sintesi organica. In particolare, l’unità dell’IBB effettuerà misure di affinità di legame, saggi di inibizione e studi cristallografici. Più in dettaglio, i target virali NSP10, NSP13, NSP14 saranno clonati, espressi in E. coli e purificati. Sarà valutata l’affinità delle molecole identificate e sintetizzate dagli altri due partner del progetto verso questi target e verso gli RNA virali G4 mediante diverse tecniche biofisiche come la termoforesi su microscala e/o la spettroscopia di fluorescenza. Inoltre, la calorimetria a titolazione isotermica sarà utilizzata per determinare anche la variazione di entalpia e la stechiometria di legame nell’interazione tra i ligandi sintetizzati con le proteine target e i G4 di RNA virale. I ligandi migliori emersi da queste valutazioni saranno co-cristallizzati con le proteine/RNA target per comprendere i dettagli molecolari responsabili dell’interazione e fornire una base strutturale per un’ulteriore ottimizzazione.
Insieme agli altri partner, l’unità dell’IBB sarà coinvolta nella diffusione dei risultati del progetto e coordinerà la gestione del progetto.
Risultati attesi:
• Costanti di dissociazione e inibizione dei composti sintetizzati contro i loro target virali;
• Determinazione della stechiometria di legame e dei parametri termodinamici correlati ai processi di associazione;
• Struttura cristallografica dei complessi target virale/ligando.
Proponenti
- Istituto di Biostrutture e Bioimmagini-CNR
Enti coinvolti
- Università degli studi di Napoli “Federico II”
- Università “Magna Grecia” di Catanzaro
Responsabile scientifico
Dr. Giuseppina De Simone (PI)

